| 产品名称:3D 细胞培养和细胞生长限制-创新 3D 细胞培养系统的新进展 |
| 品牌: |
| 货号: |
| 价格:询价 |
| 联系人:陈经理 |
| 电话:18618101725 |
3D 细胞培养和细胞生长限制-创新 3D 细胞培养系统的新进展
> 摘要 本综述概述了传统的和正在发展中的生物细胞培养技术,重点介绍了 3D 细胞培养技术相对于 2D 细胞培养技术的优势。通过将微流控技术与细胞研究相结合,可以进一步实现这些优势,从而对关键的细胞培养参数(例如细胞微环境的空间和时间梯度)进行前所未有的控制。细胞限制、针对性地分离和变形少量细胞是微流控技术实现的一项新技术,进一步扩展了微流控技术的应用和潜力,而这些应用和潜力尚未在该研究领域得到充分实现。
> 简介 为了充分了解活细胞的复杂形态和功能,必须考虑细胞的高度动态特性,例如其三维结构、机械性能和生化环境 [1]。 体外细胞培养技术长期以来一直是重要的研究和工业工具 [2],用于通过从癌症药物筛选到发育生物学的众多研究来更好地了解细胞行为 [3]。传统上,细胞培养基于二维玻璃、聚苯乙烯或硬塑料基质;这些基于“培养皿”的细培养物产生的细胞单层因无法重建体内结构而受到严重限制。由于二维细胞培养物与细胞微环境不同,因此不能代表生物体内的生物反应[1], [4]。因此,二维细胞培养研究中测量的生物反应(如受体表达、细胞迁移和细胞凋亡)可能与细胞原始组织或环境中的生物反应有显著差异[3]。
三维细胞培养的出现是为了解决二维细胞培养的局限性 [1]。这些 3D 细胞培养用于模拟天然细胞外基质 (ECM),这是一种高度水合的胶原蛋白和弹性纤维网络,嵌入由糖胺聚糖、蛋白聚糖和糖蛋白组成的凝胶状物质中[5]。它们可以使用天然和合成材料以基质或支架的形式制造,以更好地代表细胞环境的空间组织[3],许多细胞的行为都依赖于此,从而缩小了细胞培养与天然生物环境之间的差距[4],[6]。因此,3D细胞培养已成为提高细胞研究相关性的重要手段[7]。
近几十年来,微流控技术的出现进一步扩展了我们改进细胞培养的能力,让我们能够更地控制局部细胞微环境[8], [9]。微流控技术是一门涉及在微米级几何形状中操纵少量流体的领域,通常在硅基芯片上进行,它为细胞研究带来了许多好处,例如对微环境进行更好的空间和时间控制 [8]、能够使用更少的试剂量、缩短反应时间[10],并且具有更大的细胞处理集成潜力[9], [11]。微流控技术还产生了细胞限制潜力,通过这种方式,少量细胞,甚至单个细胞,都可以机械变形[12]或简单地分离[13],可以研究细胞形状和机械影响的影响,并更有针对性地了解细胞行为[14]。
本综述旨在总结 3D 细胞培养和细胞限制的传统和微流体方法,以及它们在细胞研究进展中的意义。
> 基于基质的 3D 细胞培养平台 大量研究表明,细胞微环境影响着指导细胞表型的复杂空间和时间信号传导域,因此,二维细胞培养与细胞自然环境之间的差异问题变得显而易见,其中,细胞不仅由其基因组定义,还受到许多其他影响,包括 ECM、生长因子、激素和其他小分子 [15]。
3D 细胞基质或支架是一种多孔基质,旨在支持细胞在其结构上或内部生长、增殖和分化,目的是模仿天然 ECM 特征,使基质中的细胞表现得像 在体内一样 [5]。细胞基质应用可分为临床建模或体外方法。临床方法,如组织工程或再生医学,旨在使用人工 3D 基质修复、维持或改善有缺陷的人体组织 [16]。在这种情况下,必须考虑机械和生物降解性,以便充分保护结构形状和接种细胞在植入后免受负荷,并确保支架材料可以在体内代谢而不会产生严重的毒性作用 [17]。为体外3D设计的支架建模的目的是对细胞生物学进行系统分析,以促进对细胞生理学和病理生理学的理解 [18]。在这种实验背景下,3D基质的设计应模仿实验者试图模拟的任何细胞微环境的 3D 组织和功能,并进一步注意基质对成像和分析工具的可访问性[5]。
临床和体外细胞基质应用中共同的关键设计方面是基质的宏观和微观结构,包括基质孔径和几何形状、孔隙率、孔隙的连通性和表面拓扑结构 [19]。这些多孔结构的高表面积允许细胞向内生长和固定,以及液体和营养物质的运输。根据所培养的细胞或组织的类型,支架的微孔隙率对于毛细血管生长和细胞与基质之间的相互作用很重要,而结构的大孔隙率对于营养物质和细胞代谢废物的运输重要 [3]。
细胞基质可用天然和合成材料制造,通常采用水凝胶的形式,水凝胶是一种含水量高的交联网络,在细胞培养中表现出很高的功效 [15]。天然聚合物材料,如胶原蛋白、丝素蛋白、壳聚糖、海藻酸盐和淀粉,是用于细胞培养的批生物材料 [21],通常表现出良好的生物相容性和生物降解性,尽管不同批次之间的差异和某些聚合物的加工难度是显著的缺点 [19],[22]。合成聚合物,如聚乙醇酸 (PLG) 和聚乳酸 (PLA),虽然生物相容性通常不如天然聚合物,但为更好地控制关键特性提供了机会,如表面积与体积之比、孔隙率和机械性能 [19],[22]。尽管 3D 细胞基质为细胞培养技术带来了特的优势,但它们仍然无法完实现化学物质和氧气的时空梯度或机械活性微环境,而这些正是细胞环境的关键特征 [1]。
> 微流控3D细胞培养平台 微流控技术与细胞生物学的结合,使人们能够前所未有地控制决定细胞行为的细胞微环境中化学物质和氧气的空间和时间梯度,从而能够在微尺度上调整变量,而这是传统细胞培养模型无法实现的 [8], [9]。这些专为细胞生物学应用而设计的微流控设备将 3D 细胞培养分析与微芯片行业的微制造技术相结合 [1]。聚二甲基硅氧烷 (PDMS) 是一种硅基材料,是微流控细胞培养研究中受欢迎的培养基,通常使用软光刻技术制成具有微尺度通道的小芯片 [9]。可以通过使用微流控网络运输化学物质、营养物质,实现对细胞微环境的控制,和氧气以用户确定的流速流动,并通过操纵芯片的微加工和微图案化过程来控制细胞-ECM 相互作用和施加在细胞上的剪切力 [11]。微流体方法还带来了实际好处,能够处理明显更小的样本,从而有可能降低细胞研究的成本,同时允许将分析标准操作轻松集成到系统中 [14],[23]。有可能降低细胞研究的成本,同时可以轻松地将分析标准操作集成到系统中 [14], [23]。有可能降低细胞研究的成本,同时可以轻松地将分析标准操作集成到系统中 [14], [23]。
虽然该领域的大部分进展都是与不同微流控几何形状和平台相关的概念验证演示 [9],[23],但已经开发了许多微流控系统,包括那些应用于生物传感器开发、药物筛选、干细胞研究和基因分析的系统 [11]。微流控 3D 细胞培养的进展也体现在 ECM 建模中,用于支持 3D 细胞生长和检查细胞迁移,这是活细胞的重要特性,影响胚胎发生、伤口愈合、免疫反应、组织发育和疾病过程,如癌症转移和炎症 [24]–[27]。这些微流控细胞迁移试验已被证明可以用有限的试剂产生准确的细胞迁移行为,同时克服了传统二维细胞迁移试验的一些缺点,例如通过细胞刮擦方法可能对细胞造成损害,以及此类模型不适合扩大到高通量筛选[24]。
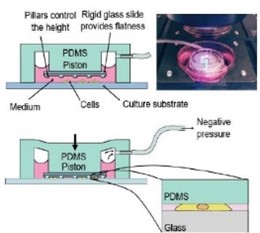
> 细胞限制 微流控与细胞生物学的结合带来了一种新功能,即所谓的细胞限制,包括单个细胞的机械变形和细胞分离。微流控装置已用于将单个细胞分离在纳升和皮升大小的液滴中,从而大大提高了细胞浓度。这使得细胞释放的分子可以更快地积累,而无需预孵育阶段,从而可以快速检测细菌[13],[29]。
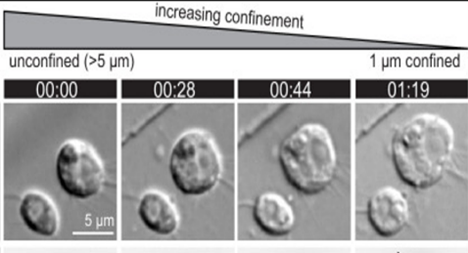
细胞限制还可用于将细胞强制进入特定的几何形状(即弯曲的微流体通道),从而将所需的形状施加到细胞上,从而可以研究细胞形状和机械应力对许多特性的影响 [30]。细胞限制已用于许多不同的细胞应用,例如,研究细胞骨架与细胞性之间的关系 [31]、限制下的细胞迁移行为 [32]、癌细胞机械特性 [12]、细胞分离 [33],以及基于其可变形性对特定细胞进行无标记识别 [34]。这些研究中使用的细胞限制技术各不相同,从利用特的微流体通道几何形状 [31] [35] 到光学拉伸器 [12] 和惯性力 [34],展示微流体技术应用于细胞生物学的多功能性。 法国4DCell公司Dynamic Cell Confiner-将细胞控制在两个平面之间,对单层细胞进行动态定量压缩刺激
Static Cell Confiner-将细胞控制在两个平面之间,对单层细胞进行静态定量压缩刺激
产品图片由其中国代理商世联博研(北京)科技有限公司提供
> 结论 总之,与传统的二维细胞培养相比,三维细胞培养具有广泛的优势。 体内细胞自然存在的空间域对于决定细胞行为的生物信号传导至关重要,但无法在二维细胞培养模型中产生的细胞单层上可靠地实现。虽然目前的三维细胞培养技术,即结合细胞基质或支架的技术,大大改进了二维细胞培养技术,但它们仍然缺乏复制细胞微环境关键方面的能力。其中一些方面,包括化学物质和氧气的空间和时间梯度以及机械刺激,可以通过使用微流体技术来解决,微流体技术与细胞生物学相结合后,可提供具有高度通用应用的细胞培养平台。这包括细胞限制的新颖能力,有针对性地洞察细胞变形和机械应力的影响。尽管许多细胞生物学微流体研究只是示范 [9],但很明显,通过利用微流体,细胞行为研究将具有越来越大的潜力,而且越来越有影响力和实用性。
> 参考文献 [1] D. Huh, G. A. Hamilton, and D. E. Ingber, “From 3D cell culture to organs-on-chips,” Trends Cell Biol., vol. 21, no. 12, pp. 745–754, 2011. [2] M. Ravi, V. Paramesh, S. R. Kaviya, E. Anuradha, and F. D. Paul Solomon, “3D cell culture systems: Advantages and applications,” J. Cell. Physiol., vol. 230, no. 1, pp. 16–26, 2015. [3] J. W. Haycock, 3D cell culture: a review of current approaches and techniques., vol. 695. 2011. [4] F. Pampaloni, E. G. Reynaud, and E. H. K. Stelzer, “The third dimension bridges the gap between cell culture and live tissue.,” Nat. Rev. Mol. Cell Biol., vol. 8, no. 10, pp. 839–845, 2007. [5] J. Lee, M. J. Cuddihy, and N. A. Kotov, “Three-dimensional cell culture matrices: state of the art.,” Tissue Eng Part B Rev, vol. 14, no. 1, pp. 61–86, 2008. [6] M. Vinci et al., “Advances in establishment and analysis of three-dimensional tumor spheroid-based functional assays for target validation and drug evaluation,” BMC Biol., vol. 10, no. 1, p. 29, 2012. [7] B. A. Justice, N. A. Badr, and R. A. Felder, “3D cell culture opens new dimensions in cell-based assays,” Drug Discov. Today, vol. 14, no. 1–2, pp. 102–107, 2009. [8] I. Meyvantsson and D. J. Beebe, “Cell culture models in microfluidic systems.,” Annu. Rev. Anal. Chem., vol. 1, pp. 423–449, 2008. [9] E. W. K. Young and D. J. Beebe, “Fundamentals of microfluidic cell culture in controlled microenvironments,” Chem Soc Rev, vol. 39, no. 3, pp. 1036–1048, 2010. [10] D. J. Beebe, G. a Mensing, and G. M. Walker, “Physics and applications of microfluidics in biology.,” Annu. Rev. Biomed. Eng., vol. 4, pp. 261–286, 2002. [11] J. El-Ali, P. K. Sorger, and K. F. Jensen, “Cells on chips.,” Nature, vol. 442, no. 7101, pp. 403–411, 2006. [12] J. Guck et al., “Optical deformability as an inherent cell marker for testing malignant transformation and metastatic competence,” Biophys J, vol. 88, no. 5, pp. 3689–3698, 2005. [13] S. K?ster et al., “Drop-based microfluidic devices for encapsulation of single cells.,” Lab Chip, vol. 8, no. 7, pp. 1110–1115, 2008. [14] H. Andersson and A. Van den Berg, “Microfluidic devices for cellomics: A review,” Sensors Actuators, B Chem., vol. 92, no. 3, pp. 315–325, 2003. [15] M. W. Tibbitt and K. S. Anseth, “Hydrogels as extracellular matrix mimics for 3D cell culture,” Biotechnol. Bioeng., vol. 103, no. 4, pp. 655–663, 2009. [16] J. P. Vacanti and R. Langer, “Tissue engineering: the design and fabrication of living replacement devices for surgical reconstruction and transplantation.,” Lancet, vol. 354, p. SI32-I34, 1999. [17] G. S. D. Hetal Patel, Minal Bonde, “Biodegradable polymer scaffolds for tissue engineering,” Trends Biomater. Artif. Organs, vol. 25, no. 1, pp. 20–29, 2011. [18] L. G. Griffith and M. A. Swartz, “Capturing complex 3D tissue physiology in vitro.,” Nat. Rev. Mol. cell Biol., vol. 7, no. 3, pp. 211–24, 2006. [19] D. J. Tobin, “Scaffolds for Tissue Engineering and 3D Cell Culture,” Methods Mol. Biol., vol. 695, no. 2, pp. 213–227, 2011. [20] J. Naranda et al., “Polyester type polyHIPE scaffolds with an interconnected porous structure for cartilage regeneration,” Sci. Rep., vol. 6, no. February, p. 28695, 2016. [21] B. Dhandayuthapani, Y. Yoshida, T. Maekawa, and D. S. Kumar, “Polymeric scaffolds in tissue engineering application: A review,” Int. J. Polym. Sci., vol. 2011, no. ii, 2011. [22] F. J. O’Brien, “Biomaterials & scaffolds for tissue engineering,” Mater. Today, vol. 14, no. 3, pp. 88–95, 2011. [23] A. L. Paguirigan and D. J. Beebe, “Microfluidics meet cell biology: Bridging the gap by validation and application of microscale techniques for cell biological assays,” BioEssays, vol. 30, no. 9, pp. 811–821, Sep. 2008. [24] F.-Q. Nie, M. Yamada, J. Kobayashi, M. Yamato, A. Kikuchi, and T. Okano, “On-chip cell migration assay using microfluidic channels.,” Biomaterials, vol. 28, no. 27, pp. 4017–4022, 2007. [25] A. Valster, N. L. Tran, M. Nakada, M. E. Berens, A. Y. Chan, and M. Symons, “Cell migration and invasion assays,” Methods, vol. 37, no. 2, pp. 208–215, 2005. [26] C. R. Justus, N. Leffler, M. Ruiz-Echevarria, and L. V Yang, “In vitro cell migration and invasion assays.,” J. Vis. Exp., vol. 752, no. 88, p. e51046, 2014. [27] N. Kramer et al., “In vitro cell migration and invasion assays.,” Mutat Res, vol. 752, no. 1, pp. 10–24, 2013. [28] J. W. Hong, V. Studer, G. Hang, W. F. Anderson, and S. R. Quake, “A nanoliter-scale nucleic acid processor with parallel architecture.,” Nat. Biotechnol., vol. 22, no. 4, pp. 435–439, 2004. [29] J. Q. Boedicker, L. Li, T. R. Kline, and R. F. Ismagilov, “Detecting bacteria and determining their susceptibility to antibiotics by stochastic confinement in nanoliter droplets using plug-based microfluidics.,” Lab Chip, vol. 8, no. 8, pp. 1265–1272, 2008. [30] G. Velve-Casquillas, M. Le Berre, M. Piel, and P. T. Tran, “Microfluidic tools for cell biological research,” Nano Today, vol. 5, no. 1. pp. 28–47, 2010. [31] C. R. Terenna et al., “Physical Mechanisms Redirecting Cell Polarity and Cell Shape in Fission Yeast,” Curr. Biol., vol. 18, no. 22, pp. 1748–1753, Nov. 2008. [32] G. Faure-andré, “Regulation of Dendritic Cell Migration by CD74, the MHC Class II–Associated Invariant Chain,” Science (80-. )., vol. 1705, no. December, 2008. [33] S. M. McFaul, B. K. Lin, and H. Ma, “Cell separation based on size and deformability using microfluidic funnel ratchets,” Lab Chip, vol. 12, no. 13, pp. 2369–2376, 2012. [34] S. C. Hur, N. K. Henderson-MacLennan, E. R. B. McCabe, and D. Di Carlo, “Deformability-based cell classification and enrichment using inertial microfluidics.,” Lab Chip, vol. 11, no. 5, pp. 912–920, 2011. [35] H. W. Hou, Q. S. Li, G. Y. H. Lee, A. P. Kumar, C. N. Ong, and C. T. Lim, “Deformability study of breast cancer cells using microfluidics,” Biomed. Microdevices, vol. 11, no. 3, pp. 557–564, 2009. |